- 23 Oct, 2023
- read
- Karolinehc
Categories: Visualización de Datos Proyectos con Python
Tags: Plotly COVID-19 Python Mapas Coropléticos Pandas Datos de Salud Gráficos Interactivos Estadísticas Globales
El contenido presentado en este artículo está destinado únicamente para fines académicos. Las opiniones expresadas se basan en mi comprensión e investigación personal. Es importante tener en cuenta que el campo de los grandes datos y los lenguajes de programación discutidos, como Python, R, Power BI, Tableau y SQL, son dinámicos y están en constante evolución. Este artículo tiene como objetivo fomentar el aprendizaje, la exploración y la discusión dentro del campo en lugar de proporcionar respuestas definitivas. Se recomienda la discreción del lector.
En los últimos tiempos, nuestro mundo se ha visto profundamente afectado por la pandemia de COVID-19 Who WHO, una crisis de salud global que trajo desafíos y cambios sin precedentes a nuestra vida diaria. La pandemia puso de relieve la importancia del análisis y la visualización de datos para comprender la propagación del virus, su impacto en diferentes regiones y la eficacia de las medidas de salud pública. Una herramienta poderosa para visualizar dichos datos es Plotly, una biblioteca de Python que nos permite crear diagramas y gráficos interactivos e informativos. En este contexto, exploraremos cómo Plotly PLOTLY puede ayudarnos a obtener información sobre la situación de COVID-19 en constante evolución.
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
from statsmodels.formula.api import ols
import plotly.graph_objects as go
import plotly.express as px
covid0 = pd.read_csv(r'D:\helen\Documents\PythonScripts\datasets\turi\covid_data.csv')
covid0.head()
En este caso, Afganistán tenía una población estimada de alrededor de 39 millones de personas al 31 de diciembre de 2019.
por cada 1.000 personas que vivían en Afganistán, había, en promedio, la mitad de una cama de hospital disponible para su uso
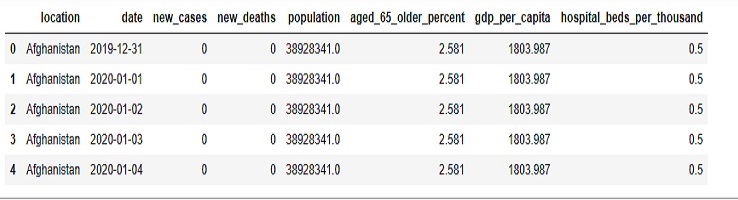
NAN = [(c,covid0[c].isna().mean()*100) for c in covid0]
NAN = pd.DataFrame(NAN, columns=['column_name', 'percentage'])
NAN
No hay valores o perdida de data
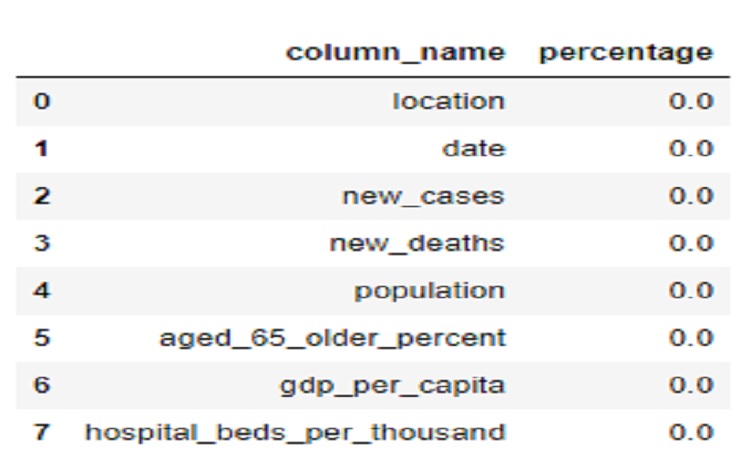
covid0.describe()
Estadísticas: la diferencia entre la media y el percentil 50 (mediana) de la columna “población” es significativa.
La población media es de aproximadamente 103.700.300 (1.037003e+08), que es la población promedio en todos los puntos de datos del conjunto de datos.
La población del percentil 50 (mediana) es de aproximadamente 9.449.321. Este valor representa el punto medio de los datos de población cuando se organizan en orden ascendente. No se ve influenciado por valores extremadamente altos o bajos.
La diferencia entre estos dos valores muestra que la población media es significativamente mayor que la población mediana. Esto sugiere que hay algunos países con poblaciones muy grandes que están sesgando la media, haciéndola mucho más alta que la población de la mayoría de los países. La mediana, por otro lado, nos da una idea de la población “típica”, que es mucho menor que la media debido a la influencia de los pocos países con poblaciones muy altas.
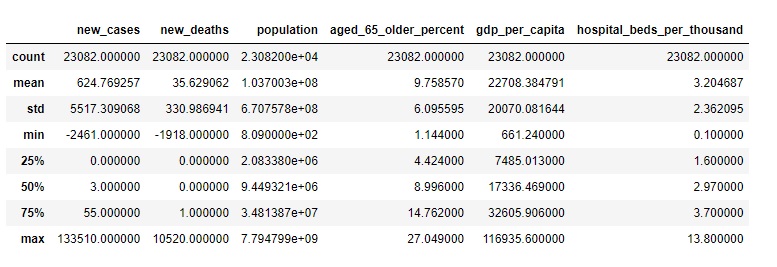
covid0[covid0['location']=='Colombia']
Para el caso colombiano, al 6 de junio de 2020 se reportaron 1.515 nuevos casos de COVID-19 y 58 nuevas muertes. En cuanto a descripciones adicionales:
Población: La población estimada de Colombia en ese momento era de aproximadamente 50.882.884 personas.
Porcentaje de 65 años o más: El porcentaje de la población de 65 años o más fue del 7,646%, lo que indica que alrededor del 7,646% de la población estaba dentro del grupo de edad de 65 años o más.
Camas de hospital por cada mil: Había aproximadamente 1,71 camas de hospital disponibles por cada 1.000 habitantes en Colombia, lo que significa que había alrededor de 1,71 camas de hospital por cada 1.000 personas que vivían en Colombia.
fig = go.Figure(go.Bar(
x=covid0_goon['date'],
y=covid0_goon['new_cases'],
))
fig.update_layout(
title='Cases day by day',
template='plotly_white',
xaxis_title='Cases',
yaxis_title='Days',
)
fig.show()
Este código crea un gráfico de barras que visualiza los datos de ’nuevos_casos’ a lo largo del tiempo, con la fecha en el eje x y el número de casos nuevos en el eje y. También establece títulos y la plantilla de trazado para una visualización limpia e informativa.
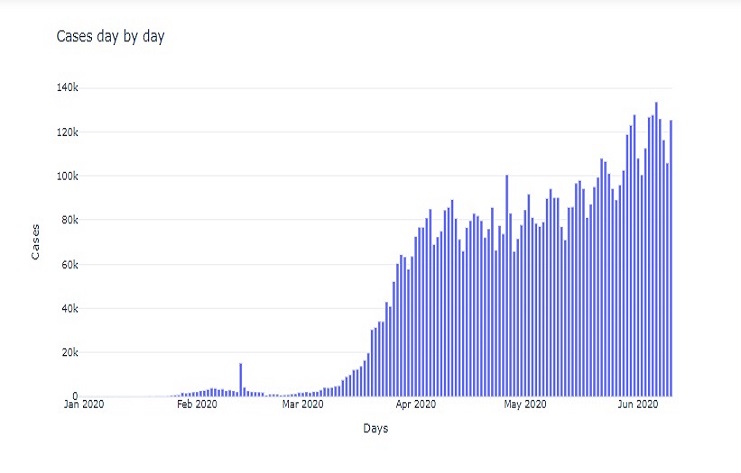
fig = px.choropleth(covid0_countries, locations=covid0_countries['Country'],
color=covid0_countries['Total Cases'],locationmode='country names',
hover_name=covid0_countries['Country'],
color_continuous_scale=px.colors.sequential.OrRd, template='plotly_dark',)
fig.update_layout(
title='Cases per Country',
)
fig.show()
El resultado es un mapa coroplético que visualiza el número de casos de COVID-19 por país, utilizando colores para representar los valores de “Casos totales” para cada país. El mapa nos permite pasar el cursor sobre los países para ver información adicional y se presenta con un fondo oscuro para una mejor visibilidad.
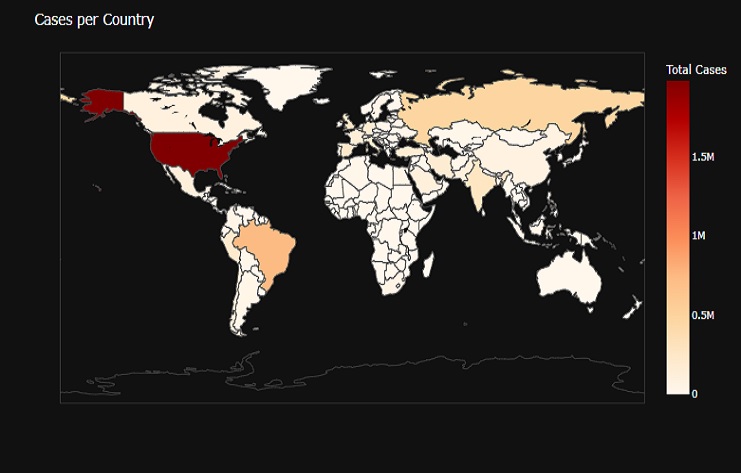
fig = px.choropleth(covid0_countries_pop, locations=covid0_countries_pop['location'],
color=covid0_countries_pop['Beds Per Thousand'], locationmode='country names',
hover_name=covid0_countries_pop['location'],
color_continuous_scale=px.colors.sequential.OrRd, template='plotly_dark',)
fig.update_layout(
title='Beds per Country',
)
fig.show()
El resultado es un mapa coroplético que visualiza el número de camas de hospital por cada mil habitantes en cada país. El color de cada país se basa en los valores de ‘Camas por mil’. Al pasar el cursor sobre un país se muestra su nombre y el mapa se presenta con un fondo oscuro para una mejor visibilidad.
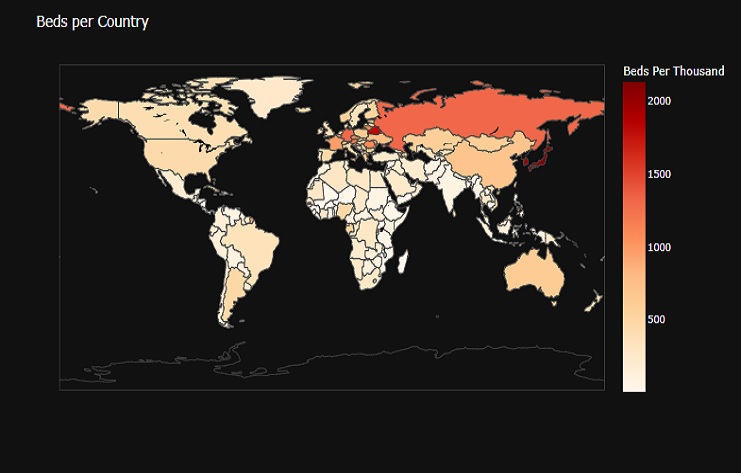
fig = go.Figure()
fig.add_trace(go.Scatter(x = covid0_co['date'], y = covid0_co['new_cases'],
mode='lines',
name='Cases in Colombia'))
fig.add_trace(go.Scatter(x = covid0_bz['date'], y = covid0_bz['new_cases'],
mode='lines',
name='Cases in Brazil'))
fig.update_layout(
title='Cases in South America',
template='plotly_white'
)
fig.show()
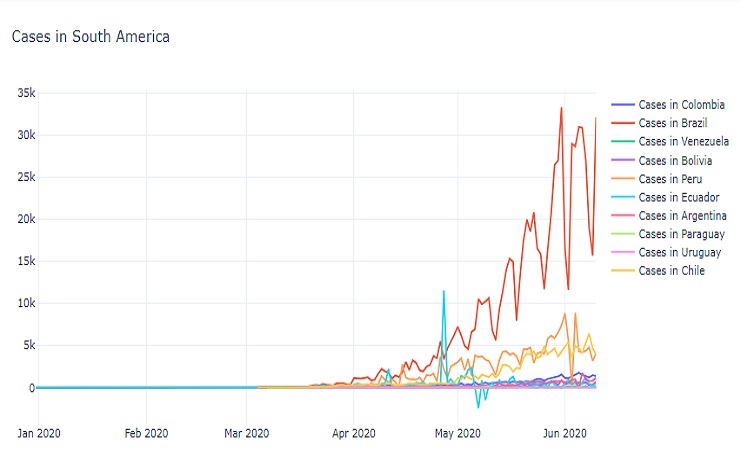
El resultado es un gráfico de líneas que muestra los nuevos casos diarios de COVID-19 en varios países de América del Sur y una ubicación adicional a lo largo del tiempo. El gráfico proporciona información sobre las tendencias de los nuevos casos en estas regiones.